Los genomas de referencia a nivel cromosómico sustentan el estudio de la genómica funcional, comparativa y poblacional dentro y entre especies. Los primeros conjuntos de genomas de alta calidad de humanos 1 y otras especies modelo (por ejemplo, Caenorhabditis elegans 2 , ratón 3 y pez cebra 4 ) se reunieron utilizando lecturas de secuenciación de Sanger de 500 a 1000 pares de bases (pb) de millas de lecturas organizadas jerárquicamente clones con inserciones de 200-300 kilobase (kb) y mapas genéticos de cromosomas. Este enfoque requirió un gran esfuerzo manual, ingeniería de software y costos, en proyectos de una década.Los enfoques de escopeta de genoma completo simplificaron la logística (por ejemplo, en humanos 7 y Drosophila 8), y la secuenciación posterior de próxima generación con lecturas de secuenciación más cortas (30-150 pb) y tamaños de inserto cortos (por ejemplo, 1 kb) marcó el comienzo de una secuenciación del genoma más asequible y escalable 9 . Sin embargo, las lecturas más cortas dieron como resultado ensamblajes de menor calidad, fragmentados en millas de piezas, donde faltaban muchos genes, estaban truncados o ensamblados incorrectamente, lo que resultóba en anotaciones y otros errores 10 Tales errores pueden requerir meses de esfuerzo manual para corregir genes individuales y años para corregir un ensamblaje completo. .La heterocigosidad genómica planteó problemas adicionales, porque los haplotipos homólogos en un genoma diploide o poliploide son forzados a unirse en un único consenso por ensambladores estándar, creando a veces duplicaciones de genes falsas 11 , 12 , 13 , 14 .
Para abordar estos problemas, el consorcio G10K 5 , 6 inició el Proyecto de Genomas de Vertebrados (VGP; https://vertebrategenomesproject.org ) con el objetivo final de producir al menos un cromosoma de alta calidad, casi libre de errores y sin espacios . , ensamblaje del genoma de referencia anotado y en fase de haplotipos para cada una de las 71.657 especies de vertebrados nombradas existentes y el uso de estos genomas para abordar cuestiones fundamentales en biología, enfermedades y conservación de la biodiversidad. Con este fin, habiendo aprendido las lecciones de tener demasiadas variables que hacen que las conclusiones sean más difíciles de alcanzar en el G10K del esfuerzo del G10K Assemblathon 2 15, primero evaluamos enfoques de secuenciación y ensamblaje del genoma extensamente en una especie, el colibrí de Anna ( Calypte anna ). Luego implementamos el método de mejor rendimiento en dieciséis especies que representan seis clases principales de vertebrados, con una amplia diversidad de características genómicas. Basándonos en los principios aprendidos, mejoramos aún más estos métodos, descubrimos parámetros y enfoques que funcionan mejor para especies con diferentes características genómicas y realizamos descubrimientos biológicos que no han sido posibles con los ensamblajes anteriores.
Los ensamblajes completos y precisos requieren lecturas largas
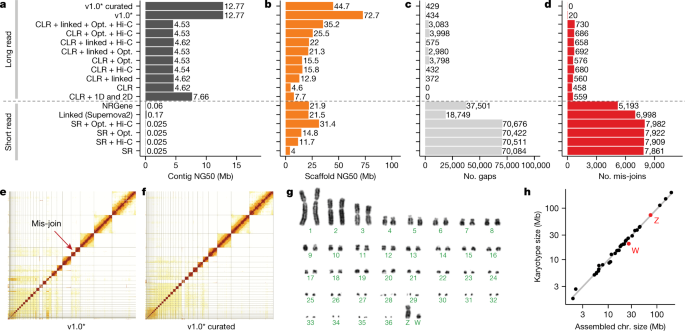
Elegimos una hembra de colibrí de Anna porque tiene un genoma relativamente pequeño (alrededor de 1 Gb), es heterogamético (tiene cromosomas sexuales Z y W) y tiene una referencia anotada del mismo individuo construida a partir de lecturas cortas 16 . Obtuvimos 12 nuevos tipos de datos de secuenciación, incluidas lecturas cortas y largas (80 bp a 100 kb) e información de enlace de largo alcance (40 kb a más de 100 Mb), generada utilizando ocho tecnologías (Tabla complementaria 1 ). Comparamos todas las tecnologías y algoritmos de ensamblaje (Tabla complementaria 2 ) de forma aislada y en muchas combinaciones (Tabla complementaria 3). Hasta donde sabemos, este fue el primer análisis sistemático de muchas tecnologías de secuencia, algoritmos de ensamblaje y parámetros de ensamblaje aplicados al mismo individuo. Encontramos que las secuencias contiguas primarias (contigs) (pseudohaplotipo; Nota complementaria 1 ) ensambladas a partir de lecturas largas continuas (CLR) de Pacific Biosciences o lecturas largas de Oxford Nanopore (ONT) eran aproximadamente de 30 a 300 veces más largas que las ensambladas de Illumina lecturas cortas (SR), independientemente de la combinación de tipos de datos o el algoritmo de ensamblaje utilizado (Fig. 1a , Tabla complementaria 3 ). Los contig NG50 más altos para ensamblajes de solo lectura cortos fueron de aproximadamente 0.025 a 0.169 Mb, mientras que para lecturas largas fueron de aproximadamente 4.6 a 7.66 Mb (Fig. 1a); contig NG50 es una métrica de ensamblaje basada en una mediana ponderada de las longitudes de sus secuencias sin espacios en relación con el tamaño estimado del genoma. Después de fijar una función en el software PacBio FALCON 17 que causaba roturas artificiales en contigs entre tramos de secuencias de haplotipos altamente homocigotos y heterozigotos (Nota complementaria 1 , Tabla complementaria 2 ), contig NG50 casi se triplicó a 12,77 Mb (Fig. 1a ). Estos hallazgos son consistentes con las predicciones teóricas 18 y demostrar que, dada la tecnología de secuenciación actual y los algoritmos de ensamblaje, no es posible lograr una alta continuidad de contig solo con lecturas cortas, ya que normalmente es imposible hacer un puente a través de repeticiones que son más largas que la longitud de lectura.
a , Valores de Contig NG50 del pseudohaplotipo primario. b , Valores del andamio NG50. c , Número de uniones (huecos). d , Número de errores de unión incorrecta en comparación con el ensamblaje seleccionado. El ensamblado curado no tiene conflictos restantes con los datos sin procesar y, por lo tanto, no se conocen errores de combinación. * Igual que CLR + vinculado + Opc. + Hi-C, pero con contigs generados con una versión actualizada de FALCON 17 y una versión anterior de Hi-C Salsa (v2.0 versus v2.2; Tabla complementaria 2 ) para uniones de contig menos agresivas. e , f, Mapas de calor de interacción Hi-C antes y después de la curación manual, que identificaron 34 cromosomas. Las líneas de cuadrícula indican los límites del andamio. Flecha roja, ejemplo de combinación incorrecta que se corrigió durante la curación. g , Cariotipo de los cromosomas identificados ( n = 36 + ZW), consistente con hallazgos previos 70 . h , Correlación entre los tamaños de cromosomas estimados (en Mb) basados en imágenes de cariotipo en gy andamios ensamblados en la Tabla complementaria 4 (bCalAna1) en una escala logarítmica. v1.0, tubería de ensamblaje VGP v1.0; lecturas vinculadas 10X Genomics; Ligadura de proximidad Hi-C, Hi-C; Lecturas largas 1D, 2D, Oxford Nanopore; Lecturas de Illumina de extremo emparejado NRGene, NRGene; SR, lecturas cortas de Illumina de extremo emparejado.
Tubería de montaje iterativa
Los andamios generados con las tres tecnologías de andamios (es decir, lecturas vinculadas 10X Genomics (10XG), mapas ópticos Bionano (Opt.) Y Arima Genomics, Dovetail Genomics o Phase Genomics Hi-C) fueron aproximadamente 50% a 150% más largos que los generados usando una o dos tecnologías, independientemente de si comenzamos con contigs de lectura corta o larga (Fig. 1b , Datos extendidos Fig. 1a , Tabla complementaria 3 ). Estos hallazgos incluyen mejoras que hicimos en cada enfoque (Nota complementaria 1 , Tablas complementarias 4 , 5 , Figura complementaria 1). A pesar de una continuidad similar del andamio, los conjuntos de sólo lectura corta tenían entre 18.000 y 70.000 espacios, mientras que los conjuntos de lectura larga tenían sustancialmente menos espacios (entre 400 y 4.000) (figura 1c ). Muchas lagunas en los ensamblajes de lectura corta estaban en regiones repetidas o ricas en GC. Teniendo en cuenta que la versión seleccionada de este ensamblaje es más precisa, también identificamos aproximadamente entre 5.000 y 8.000 combinaciones incorrectas en ensamblajes de lectura corta, mientras que los ensamblajes basados en lecturas largas solo tenían entre 20 y alrededor de 700 combinaciones incorrectas (Fig. 1d ). Estas uniones erróneas incluyeron uniones e inversiones quiméricas. Después de que curamos este ensamblaje para detectar contaminación, errores de ensamblaje y asignaciones de cromosomas basados en Hi-C ( Fig.1e, f), el ensamblaje final del colibrí tenía 33 andamios que coincidían estrechamente con el cariotipo cromosómico en número (33 de 36 autosomas más cromosomas sexuales) y tamaños estimados (aproximadamente 2 a 200 Mb; Fig. 1g, h ), con solo 1 a 30 espacios por autosoma (bCalAnn1 en la Tabla complementaria 6 ). De los cinco autosomas con sólo una brecha cada uno, tres (cromosomas 14, 15, y 19) tenían soporte completo spanning por al menos dos tecnologías (bloques fiables, Extended Data Fig. 1c ; bCalAnn1 en el cuadro complementario 6 ), lo que indica que el cromosoma los contigs estaban casi completos. Sin embargo, que se estaban perdiendo largas series de repeticiones teloméricas de vertebrado dentro de 1 kb de sus extremos (Extended Data Fig. 1c ; bCalAnn1 en los cuadros suplementarios6 , 7 ).
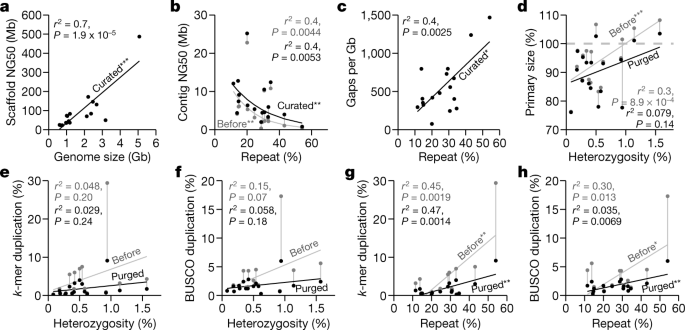
Línea de ensamblaje a través de la diversidad de vertebrados
Utilizando la fórmula que proporcionó el genoma de colibrí de la más alta calidad, construimos una tubería de ensamblaje de VGP iterativa (v1.0) con contigs CLR separados por haplotipos, seguido de andamios con lecturas vinculadas, mapas ópticos y Hi-C, y luego relleno de espacios , pulido de llamadas de base y finalmente curado manual (Datos extendidos, Figs. 2 a, 3a ). Probamos sistemáticamente nuestra línea en 15 especies adicionales que abarcan todas las clases principales de vertebrados: mamíferos, aves, reptiles no aviares, anfibios, peces teleósteos y peces cartilaginosos (Tablas complementarias 8 , 9 , Nota complementaria 2 ). Para el pinzón cebra, usamos ADN del mismo macho que se usó para generar el genoma de referencia anterior 19, e incluyó un trío de hembras para la evaluación comparativa de la completitud del haplotipo, donde las lecturas secuenciadas de los padres se utilizaron para agrupar las lecturas del haplotipo parental de la descendencia antes del ensamblaje 20 (Datos extendidos, Figuras 2 a, 3b ). Establecimos metas métricas de ensamblaje mínimo inicial de: 1 Mb contig NG50; Andamio de 10 Mb NG50; asignar el 90% de la secuencia a los cromosomas, validado estructuralmente por al menos dos líneas independientes de evidencia; Q40 calidad base media; y haplotipos ensamblados de la manera más completa y correcta posible. Cuando se lograron estas métricas, la mayoría de los genes se ensamblaron con estructuras de exones e intrones sin espacios 11y menos del 3% tenía errores de base de desplazamiento de fotogramas identificados en la anotación. Q40 es el punto de inflexión matemática en el que los genes pasan de que normalmente contienen un error a que normalmente no contienen 21 . De los ensamblajes seleccionados (Tabla complementaria 10 , Nota complementaria 2 ), 16 de 17 lograron las métricas de continuidad deseadas (Tabla de datos ampliados 1 ). El andamio NG50 se correlacionó significativamente con el tamaño del genoma (Fig. 2a ), lo que sugiere que los genomas más grandes tienden a tener cromosomas más grandes. En promedio, el 98,3% de las bases ensambladas tenían NG50 de bloque confiables que van desde 2,3 a 40,2 Mb; bases de repetición colapsadas 22con una cobertura de lectura de CLR anormalmente alta (más de 3 sd) osciló entre 0,7 y 31,4 Mb por Gb; y la integridad de los ensamblajes del genoma osciló entre 87,2 y 98,1%, con menos del 4,9% de regiones duplicadas falsamente, lo que coincide con la tasa de duplicación falsa que encontramos para el conjunto de genes de vertebrados BUSCO conservados (Tabla de datos ampliados 1 , Tablas complementarias 11 , 12 ) .
a , Correlación entre el andamio NG50 y el tamaño del genoma de los ensamblajes seleccionados. b , Correlación no lineal entre contig NG50 y contenido repetido, antes y después de la curación. c , Correlación entre el número de huecos por Gb reunidos y el contenido repetido. d , Correlación entre el tamaño del ensamblaje primario en relación con el tamaño estimado del genoma ( eje y ) y la heterocigosidad del genoma ( eje x ), antes y después de la purga de duplicaciones falsas. Los tamaños de ensamblaje superiores al 100% indican la presencia de duplicaciones falsas y los inferiores al 100% indican repeticiones colapsadas. e , f , Correlaciones entre la tasa de duplicación del genoma utilizando k -mers 23( e ) y conjunto de genes de vertebrados BUSCO conservados ( f ), y heterocigosidad del genoma antes y después de la purga de duplicaciones falsas. g , h , como en e , f , pero con contenido de repetición del genoma completo antes y después de la purga de duplicaciones falsas. El tamaño del genoma, la heterocigosidad y el contenido de repetición se estimaron a partir de recuentos de 31 meros usando GenomeScope 71 , excepto para el canal bull blenny, ya que las estimaciones no eran confiables (ver Métodos ). El contenido repetido se midió modelando la multiplicidad de k- mer a partir de lecturas de secuenciación. Las tasas de duplicación de secuencia se estimaron con Merqury 23 utilizando 21-mers. *P <0,05; ** P <0,01; *** P <0,001, del coeficiente de correlación: P valores y ajustado r 2 de F -estadísticas. n = 17 conjuntos de 16 especies.
Las repeticiones afectan notablemente la continuidad.
Para los ensamblajes generados usando nuestra tubería automatizada (Datos extendidos Fig. 3a ) antes de la curación manual, todos menos 2 (el patín espinoso y el canal toro blenny) de los 17 ensambles excedieron las métricas de continuidad deseadas (Tabla complementaria 13 ). En la búsqueda de una explicación de estos resultados, encontramos que contig NG50 disminuyó exponencialmente con el aumento del contenido de repetición, con la raya espinosa teniendo el mayor contenido de repetición (Fig. 2b , Tabla complementaria 13 ). En consecuencia, después del andamio y el llenado de huecos, observamos una correlación positiva significativa entre el contenido repetido y el número de huecos ( Fig.2c). El loro kākāpō, que tenía un 15% de contenido repetido, tenía aproximadamente 325 espacios por Gb, incluidos 2 de 26 cromosomas sin espacios (cromosomas 16 y 18) y sin evidencia de colapso o bajo soporte, lo que sugiere que los contigs cromosómicos estaban completos (bStrHab1 en la Tabla complementaria 6 ). Por el contrario, la raya espinosa, con un contenido de repetición del 54%, tenía alrededor de 1.400 huecos por Gb (Tabla de datos ampliados 1 ); ninguno de sus 49 andamios a nivel cromosómico contenía menos de ocho espacios, y todos tenían algunas regiones que contenían colapsos o poco apoyo (sAmbRad1 en la Tabla complementaria 6 ). Incluso después de la conservación y otras modificaciones para aumentar la calidad del ensamblaje (Nota complementaria 2), el número de colapsos, su tamaño total, las bases que faltan y el número de genes en los colapsos se correlacionan con el contenido repetido (Datos extendidos Fig. 4a-d ). Sin embargo, la longitud de colapso promedio se correlacionó con las longitudes de lectura de CLR promedio (10–35 kb; Datos extendidos Fig. 4e ). No hubo correlaciones entre el número de bases colapsadas y la heterocigosidad o el tamaño del genoma (Datos extendidos Fig. 4f, g ). Dependiendo de la especie, del 77,4 al 99,2% de las regiones colapsadas consistieron en duplicaciones segmentarias no resueltas (Datos extendidos, Fig. 4h ). El resto fueron repeticiones de alto número de copias, en su mayoría de tipos previamente desconocidos (Datos extendidos, Fig.4i), y de tipos conocidos como matrices de satélites, repeticiones simples, repeticiones terminales largas (LTR) y elementos nucleares intercalados cortos y largos (SINE y LINE), según la especie (Datos extendidos, Fig. 4j ). Descubrimos que el enmascaramiento repetido antes de generar contigs impedía que algunas repeticiones llegaran al ensamblaje final (Nota complementaria 3 ). Todos los hallazgos anteriores demuestran cuantitativamente el efecto que tiene el contenido repetido en la capacidad de producir ensamblajes muy continuos y completos.
Detección y eliminación de falsas duplicaciones
Durante la curación, descubrimos que uno de los errores de ensamblaje más comunes era la introducción de duplicaciones falsas, que pueden malinterpretarse como duplicaciones de exón, gen completo o grandes segmentos. Observamos dos tipos de duplicaciones falsas: 1) duplicaciones de heterotipos, que ocurrieron en regiones de mayor divergencia de secuencia entre los haplotipos paternos y maternos, donde los contigs de haplotipos separados se colocaron incorrectamente en el ensamblaje primario (Datos extendidos Fig. 5a ); y 2) duplicaciones de homotipos, que ocurrieron cerca de los límites de contig o secuencias sub-colapsadas causadas por errores de secuenciación (Datos extendidos Fig. 5b). Parecían ocurrir duplicaciones de heterotipos falsos con mayor heterocigosidad. Por ejemplo, durante la curación del genoma del pinzón cebra hembra, encontramos una secuencia heterocigótica falsamente duplicada de aproximadamente 1 Mb (Datos extendidos Fig. 6a ). Este individuo pinzón cebra tenía la mayor heterocigosidad (1,6%) en relación con todos los demás genomas (0,1-1,1%). Las duplicaciones de homotipos a menudo se producían en los límites de los contig, y tenían aproximadamente la misma longitud que la secuencia se lee (Datos extendidos Fig. 6b, c ). Identificamos y eliminamos duplicaciones falsas durante la curación usando cobertura de lectura, auto-, transcripción-, mapa óptico- y alineaciones Hi-C, y perfiles k -mer (Figura 6 de datos extendidos , Figura 2 complementaria ).
Antes de purgar las duplicaciones falsas, el tamaño del genoma del ensamblaje primario se correlacionó positivamente con el porcentaje estimado de heterocigosidad; más genomas heterocigotos tendían a tener tamaños de ensamblaje mayores que el tamaño estimado del genoma haploide (Fig. 2d ). De manera similar, la tasa de duplicación adicional en el ensamblaje primario, medida con k-mers ). Para eliminar estas falsas duplicaciones automáticamente, inicialmente usamos Purge_Haplotigs 1323 or conserved vertebrate BUSCO genes24, varied from 0.3% to 30% and trended towards correlation with heterozygosity (Fig. 2e, f, Supplementary Table 13). Apparent false gene duplication rates correlated more strongly with the overall repeat rate in the assemblies (Fig. 2g, h, which removed retained falsely duplicated contigs that were not scaffolded (Extended Data Fig. 5; VGP v1.0–1.5). Later, we developed Purge_Dups14 to remove both falsely retained contigs and end-to-end duplicated contigs within scaffolds (Extended Data Fig. 5; VGP v1.6), which reduced the amount of manual curation. After we applied these tools, the primary assembly sizes and the k-mer and BUSCO gene duplication rates were all reduced, and their correlations with heterozygosity and repeat content were also reduced or eliminated (Fig. 2d–h). These findings indicate that it is essential to properly phase haplotypes and to obtain high consensus sequence accuracy in order to prevent false duplications and associated biologically false conclusions.
Curation is needed for a high-quality reference
Each automated scaffolding method introduced tens to thousands of unique joins and breaks in contigs or scaffolds (Supplementary Table 14). Depending on species, the first scaffolding step with linked reads introduced about 50–900 joins between CLR-generated contigs. Optical maps introduced a further roughly 30–3,500 joins, followed by Hi-C with about 30–700 more joins, and each identified up to several dozen joins that were inconsistent with the previous scaffolding step. Manual curation resulted in an additional 7,262 total interventions for 19 genome assemblies or 236 interventions per Gb of sequence (Supplementary Table 15). When a genome assembly was available for the same or a closely related species, it was used to confirm putative chromosomal breakpoints or rearrangements (Supplementary Table 15). These interventions indicate that even with current state-of-the-art assembly algorithms, curation is essential for completing high-quality reference assemblies and for providing iterative feedback to improve assembly algorithms. A further description of our curation approach and analyses of VGP genomes are presented elsewhere25.
Hi-C scaffolding and cytological mapping
Most large assembled scaffolds of each species spanned entire chromosomes, as shown by the relatively clean Hi-C heat map plots across each scaffold after curation (Extended Data Fig. 7), near perfect correlation between chromosomal scaffold length and karyotypically determined chromosome length (Fig. 1h), and the presence of telomeric repeat motifs on some scaffold ends (Supplementary Table 7). In our VGP zebra finch assembly, all inferred chromosomes were consistent with previously identified linkage groups in the Sanger-based reference, except for chromosomes 1 and 1B (Extended Data Fig. 8a). Their join in the VGP assembly was supported by both single CLR reads and optical maps through the junction. We also corrected nine inversion errors and filled in large gaps at some chromosome ends. In the platypus, we identified 18 structural differences in 13 scaffolds between the VGP assembly and the previous Sanger-based reference anchored to chromosomes using fluorescence in situ hybridization (FISH) physical mapping (Extended Data Fig. 8b, Supplementary Table 16). Of these 18, all were supported with Hi-C, and seven were also supported by both CLR and optical maps in the VGP assembly. Our platypus assembly also filled in many large (approximately 1–30 Mb) gaps and corrected many inversion errors (Extended Data Fig. 8b). Furthermore, we identified seven additional chromosomes (chromosomes 30–36) in the zebra finch, and eight (chromosomes 8, 9, 14, 15, 17, 19, 21, and X4; Extended Data Fig. 8a, b) in the platypus26,27. Relative to the VGP assembly, the earlier short-read Anna’s hummingbird assembly was highly fragmented (Extended Data Fig. 8c), despite being scaffolded with seven different Illumina libraries spanning a wide range of insert sizes (0.2–20 kb). The previous climbing perch assembled chromosomes were even more fragmented and also had large gaps of missing sequence (Extended Data Fig. 8d). On average, 97% ± 3% (s.d.) of the assembled bases were assigned to chromosomes (Extended Data Table 1), compared with 76% and 32% in the prior zebra finch and platypus references, respectively. We believe the comparable or higher accuracy of Hi-C relative to genetic linkage or FISH physical mapping is due to the higher sampling rate of Hi-C pairs across the genome. Nonetheless, visual karyotyping is useful for complementary validation of chromosome count and structure28.
Trios help to resolve haplotypes
Pudimos ensamblar los contigs de pinzón cebra hembra basados en trío en andamios separados a nivel de cromosomas materno y paterno (Datos extendidos Fig. 9a ) utilizando nuestra tubería de trío VGP (Datos extendidos Fig. 3b ). En comparación con el ensamblaje no trío del mismo individuo, la versión trío tenía de siete a ocho veces menos duplicaciones falsas ( k -mer y BUSCO dups en las Tablas complementarias 11 , 12 ), variantes específicas de haplotipo bien conservadas ( precisión k -mer / recall 99.99 / 97.08%), y una mayor precisión de llamada base, excediendo Q43 para ambos haplotipos (Tabla de datos extendidos 1). El ensamblaje basado en trío fue el único ensamblaje con una separación casi perfecta (99,99%) de los haplotipos maternos y paternos, determinada utilizando k -mers específicos para cada 23 . Identificamos variantes estructurales específicas de haplotipo, incluidas inversiones de 4,5 a 12,5 Mb en los cromosomas 5, 11 y 13 que no eran fácilmente identificables en la versión sin trío (Datos extendidos Fig. 10a-e ). En el futuro, la VGP está priorizando la colección de tríos madre-padre-descendencia cuando sea posible, o dúos monoparentales-descendientes, para ayudar con el ensamblaje y la fase diploide, así como el desarrollo de métodos mejorados para el ensamblaje de genomas diploides en el ausencia de datos genómicos de los padres, como se describe en otro estudio 29 .
Efectos del pulido en la precisión
A pesar de su mayor continuidad y precisión estructural, los ensamblajes basados en CLR requirieron al menos dos rondas de pulido por consenso de lectura corta para alcanzar una precisión de nivel base del 99,99% (un error por 10 kb, Phred 30 Q40; Tabla complementaria 5 ). Antes del pulido, la precisión por base era Q30–35 (calculada utilizando k -mers). Los errores más comunes fueron indeles cortos de llamadas de consenso inexactas durante la formación de contig CLR, lo que resultó en errores de desplazamiento de marco de aminoácidos. Utilizando nuestro enfoque combinado de pulido de lectura larga y lectura corta aplicado en secuencias de haplotipos primarias y alternas juntas, pulimos del 82% al 99,7% del ensamblaje primario y aproximadamente el 91,3% del conjunto alternativo (Tabla complementaria 17). De la secuencia restante sin pulir, a veces se reconstruyó un haplotipo con una calidad sustancialmente inferior, porque la mayoría de las lecturas se alinearon con el haplotipo de mayor calidad (Datos extendidos, Fig. 11a ). Las falsas duplicaciones tuvieron efectos similares, donde la secuencia duplicada actuó como un atractor durante el mapeo de lectura. Los haplotipos en las regiones más homocigotas tendían a colapsarse con FALCON-Unzip 17 . Todos estos casos reclutaron lecturas de ambos haplotipos y, por lo tanto, causaron errores de cambio, que confirmamos en el ensamblaje basado en trío y corregimos al excluir los pares de lectura del otro haplotipo durante el pulido (Datos extendidos, Fig. 11b). Estos hallazgos indican que tanto la precisión de lectura de la secuencia como la cuidadosa separación de haplotipos son importantes para producir ensamblajes precisos.
Cromosomas sexuales y genomas mitocondriales
Los cromosomas sexuales han sido notoriamente difíciles de ensamblar, debido a su mayor divergencia con respecto a los autosomas y su alto contenido de repetición 31 . Hemos reunido con éxito los dos cromosomas sexuales (Z, W) para las tres especies de aves, el primer cromosoma W (hasta donde sabemos) para las aves vocales de aprendizaje (Extended Data Figs. 7 , 9b ), el X y / o cromosoma Y en los mamíferos placentarios (Lince de Canadá y dos especies de murciélagos), el cromosoma X en la raya espinosa, y por primera vez, hasta donde sabemos, los diez cromosomas sexuales (5X y 5Y) en el ornitorrinco 26 (Datos extendidos Fig. 9c ). La integridad y la continuidad de los cromosomas Z y W del pinzón cebra se mejoraron aún más mediante el ensamblaje basado en trío (Datos extendidos, Fig.9b ). Sin embargo, los conjuntos de cromosomas sexuales estaban aún más fragmentados que los autosomas, probablemente debido a su menor profundidad de secuenciación y alto contenido de repetición.
Los genomas mitocondriales (MT), que se espera que sean de 11 a 28 kb de tamaño 32 , se encontraron inicialmente en solo seis conjuntos (Tabla complementaria 18 ). Las lecturas sin procesar derivadas de MT estaban presentes, pero no se ensamblaron, en parte debido a los límites mínimos de longitud de lectura para el ensamblaje de contig inicial. Además, si el genoma de MT no estaba presente durante el pulido del genoma nuclear, las lecturas de MT sin procesar se sentían atraídas por las secuencias de MT nucleares (NuMT), convirtiéndolas incorrectamente en la secuencia de MT del orgánulo completo (Datos extendidos Fig. 11c ). Para abordar estos problemas, desarrollamos una tubería MT guiada por referencias e incluimos el genoma MT durante el pulido 33 (Datos extendidos, Fig. 3c; VGP v1.6). Con estas mejoras, reunimos de manera confiable 16 de 17 genomas de MT (Tabla complementaria 18 ) y descubrimos 2 kb de una expansión repetida de 83 pb dentro de la región de control en el kākāpō (Datos extendidos Fig. 9d ), y duplicaciones de genes Nad1 y trnL2 en la percha trepadora (Datos extendidos Fig. 9e ). Estas duplicaciones se verificaron utilizando lecturas CLR de una sola molécula que abarcaban las uniones de duplicación o incluso todo el genoma de MT. Su ausencia en referencias anteriores de MT 34 , 35es probable que resulte de la incapacidad de Sanger o de lecturas breves para resolver correctamente grandes duplicaciones. En otro lugar se informan más detalles sobre la tubería MT-VGP y los nuevos descubrimientos biológicos 33 .
Mejoras en la alineación y la anotación de lectura
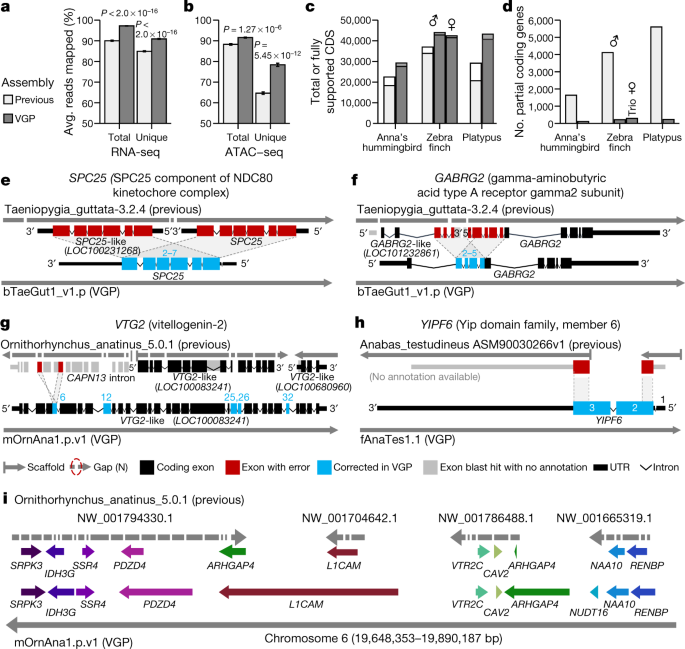
En comparación con los ensamblajes anteriores de Sanger (pinzón cebra y ornitorrinco) e Illumina (colibrí de Anna y percha trepadora), agregamos alrededor de 42-176 Mb de secuencia faltante y colocamos 68.5 Mb (pinzón cebra) a 1.8 Gb (ornitorrinco) de secuencia previamente no colocada dentro cromosomas. Corregimos alrededor de 7,800 a 64,000 combinaciones erróneas y cerramos 55,177–193,137 brechas por genoma (Tabla complementaria 19 ). De acuerdo con estas mejoras, tanto los datos de secuenciación del ARN del transcriptoma (RNA-seq) (Fig. 3a ) como el ensayo del genoma para la cromatina accesible a la transposasa utilizando datos de secuenciación (ATAC-seq) (Fig. 3b)) alineados con un 5 a 10% más de mapeo para nuestros nuevos ensambles VGP en comparación con los ensamblajes anteriores. Las anotaciones NCBI RefSeq y EBI Ensembl revelaron: 5.434 a 14.073 más transcripciones codificantes de proteínas por especie, con un 94,1 a un 97,8% de soporte completo (Fig. 3c , Tabla complementaria 20 ); sólo alrededor de 100 a 300 genes codificantes parcialmente ensamblados, en comparación con alrededor de 1.600 a 5.600 (Fig. 3d ); genes codificantes más ortólogos compartidos con humanos; y menos transcripciones que requirieran correcciones para compensar los codones de parada prematuros o los errores de indel de cambio de marco (Cuadro de datos extendido 2 ). El número total de genes anotados disminuyó en los ensamblajes de VGP (Cuadro de datos extendido 2), en parte porque hubo menos duplicaciones falsas (Tabla complementaria 19 ). Respaldando estos resultados, las asambleas de VGP tuvieron de 0 a 13% más de completitud de k- mer (95% de media ± 3,5% sd versus 88 ± 4,3%; Tabla de datos extendidos 2 , Tabla complementaria 19 ; P = 0,0047, n = 4 antes y 17 Ensambles VGP, prueba t no emparejada ).
a , b , Porcentaje promedio de muestras de transcriptoma de secuencia de ARN ( a ; n = 44, media ± sem) y lecturas de genoma de secuencia de ATAC ( b ; n = 12) que se alinean con los ensamblajes de pinzón cebra anteriores y VGP. Lecturas únicas asignadas a una sola ubicación en el ensamblaje. El total es la suma de las lecturas únicas y de múltiples mapas. Los valores de p son de la prueba t pareada . c , d , número total de transcripciones de secuencias de codificación (CDS) (barra completa) y parte totalmente compatible (barra interior) ( c ) y el número de genes codificantes de RefSeq anotados como parciales ( d) en los ensamblajes anterior y VGP utilizando los mismos datos de entrada. e – h , Ejemplos de ensamblaje y errores de anotación asociados en ensamblajes de referencia anteriores corregidos en los nuevos ensamblajes de VGP. Consulte el texto principal para obtener descripciones. i , synteny gen alrededor del VTR2C receptor en los espectáculos platypus completamente faltan genes ( NUDT16 ), truncado y duplicada ARHGAP4 , y muchas lagunas en la anterior Sanger basado en conjunto en comparación con los rellenos y expandido longitudes de genes en el nuevo VGP montaje. Las adhesiones de la asamblea se encuentran en la Tabla complementaria 19 .
Un ejemplo de una duplicación falsa de un heterotipo de gen completo en la anotación RefSeq de la referencia 19 del pinzón cebra anterior es el gen BUSCO SPC25 36 , para el cual cada haplotipo se colocó correctamente en los ensamblajes primario y alternativo de VGP (Fig. 3e ). El receptor GABRG2 , que muestra una expresión especializada en los circuitos de aprendizaje vocal 37 , tenía una duplicación parcial en tándem de cuatro de sus diez exones, lo que resultaba en duplicaciones parciales anotadas de genes en falso tándem ( similares a GABRG2 y GABRG2 ; Fig. 3f ). El gen de la vitelogenina-2 ( VTG2 ), un componente de la yema de huevo en todas las especies que ponen huevos 38, se distribuyó en 14 contigs en 3 andamios diferentes en el ensamblaje de ornitorrinco anterior (Fig. 3g ). Dos de estos andamios recibieron dos anotaciones génicas similares a VTG2 correspondientes , y el tercero se incluyó como intrón duplicado falso en CAPN-13 (rojo), que juntos causaron secuencias de aminoácidos falsas en cinco exones (azul). El gen BUSCO YIPF6 , que está asociado con la enfermedad inflamatoria intestinal 39 , se dividió entre dos andamios diferentes y, por lo tanto, se presume que es una pérdida genética en el montaje anterior de la percha trepadora 40 (Fig. 3h). Cada uno de estos genes está ahora presente en contigs VGP largos, dentro de bloques validados, sin espacios ni pérdidas o ganancias de genes falsos (Tabla complementaria 21 ).
Más allá de los genes individuales, una ventana de síntesis de diez genes que rodea al gen del receptor de vasotocina 2C ( VTR2C ; también conocido como AVPR2 ), que participa en la homeostasis de la presión arterial y la función cerebral 41 , 42 , se dividió en 34 contigs en cuatro andamios, de los cuales contenía una duplicación de haplotipos falsos de ARHGAP4 en el ensamblaje previo de ornitorrincos 43 ( Fig.3i). En nuestro ensamblaje de VGP, los once genes estaban en un contig de 37 Mb de largo dentro del andamio del cromosoma 6 de aproximadamente 50 Mb. Además, ocho de los once genes aumentaron notablemente de tamaño debido a la adición de secuencias faltantes previamente desconocidas. Esta región cromosómica era más rica en GC (54%) que todo el cromosoma 6 (46%). Miles de tales ganancias y pérdidas falsas en ensamblajes de referencia anteriores se han corregido en nuestros ensamblajes VGP (más detalles en las referencias 27 , 44 ), lo que demuestra que la calidad del ensamblaje tiene un efecto crítico en las anotaciones posteriores y la genómica funcional.
Regiones reguladoras ricas en GC de genes codificantes
Probamos si los ensamblajes VGP de mayor calidad permitían nuevos descubrimientos biológicos. En particular, comenzando alrededor de 1,5 kb corriente arriba de los genes que codifican proteínas, en bloques de 100 pb, hubo un aumento constante de aproximadamente 6-20% a aproximadamente 30-55% de genes a los que les faltaba la secuencia en referencias anteriores (Fig. 4a ); proporciones igualmente elevadas de genes carecían de sus regiones 5 ‘no traducidas (UTR) subsiguientes y de sus primeros exones. Esta fluctuación en la secuencia faltante fue directamente proporcional al contenido de GC ( Fig.4a). Por lo tanto, estudiamos el patrón de contenido de GC en todos los genes que codifican proteínas en los 16 nuevos ensamblajes de VGP y encontramos una firma en todo el genoma: un rápido aumento en el contenido de GC en aproximadamente 1,5 kb antes del sitio de inicio de la transcripción, en el 5 ′ UTR, y en el primer exón, seguido de una disminución constante en los exones posteriores y el retorno a niveles de fondo casi intergénicos en la UTR 3 ‘y aproximadamente 1,5 kb después del sitio de terminación de la transcripción (Fig. 4b ). Los intrones tenían menor contenido de GC, más cerca del fondo intergénico. El contenido de GC intergénico se mantuvo estable dentro de los 30 kb a cada lado de cada gen (Fig. 4b). Los mamíferos, aves y reptiles tuvieron el mayor aumento (alrededor del 20%) en el contenido de GC cerca del sitio de inicio, seguidos por los anfibios y las rayas con niveles medios (alrededor del 10%). Los peces teleósteos mostraron una disminución inicial, seguida de un aumento más débil (alrededor del 5%) de un contenido de GC ya más bajo (Fig. 4b ). Dado que la raya representa la rama hermana de todos los demás linajes de vertebrados secuenciados, estos hallazgos sugieren que los teleósteos perdieron al menos el 5% del contenido de GC en todo el genoma, mientras que mantuvieron la mayor parte del patrón de contenido de GC en los genes que codifican proteínas. Aunque se sabe que las regiones promotoras pueden ser ricas en CpG y el contenido de GC puede variar entre exones e intrones 45 , 46 Según nuestro conocimiento, las diferencias específicas de linaje dentro de los patrones vertebrados y la magnitud de estas diferencias no se describen en la descripción. previamente.
Fuente:
Rhie, A. (28 de abril de 2021). Hacia conjuntos genómicos completos y sin errores de todas las especies de vertebrados. Recuperado 28 de abril de 2021, de https://www.nature.com/articles/s41586-021-03451-0