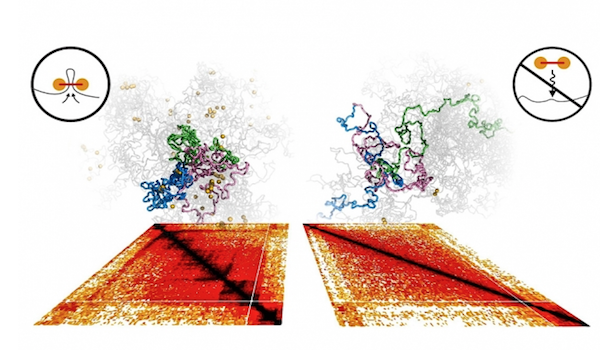
Los modelos computarizados de cromosomas y los mapas experimentales Hi-C mostrados a continuación revelan el papel central de la cohesina en doblar el genoma en dominios distintos (destacados en azul, verde y rosa). Cuando la cohesina se agota experimentalmente y en simulaciones, los dominios se despliegan y se mezclan (a la derecha).Los experimentos confirman hipótesis acerca de cómo el genoma se organiza en “barrios”.
Un motor molecular que organiza el genoma en distintos barrios formando bucles de ADN se ha caracterizado por investigadores del MIT y el Instituto Pasteur en Francia.
En un estudio publicado en 2016, un equipo liderado por Leonid Mirny, profesor de física en el Instituto de Ingeniería Médica y Ciencias del MIT, propuso que los motores moleculares transforman los cromosomas de un estado de tangos en una serie dinámica de bucles en expansión.
El proceso, conocido como extrusión de lazo, se piensa para traer los elementos reguladores junto con los genes que controlan. El equipo también sugirió que el ADN está decorado con barreras – parecidas a las señales de alto – que limitan el proceso de extrusión.
De esta manera, la extrusión de bucles divide los cromosomas en vecindarios reguladores separados, conocidos como topológicamente asociados a los dominios (TADs).
Sin embargo, mientras que los investigadores sugirieron que un anillo-como el complejo de la proteína llamado cohesin era un candidato probable para estos motores moleculares, esto todavía tenía que ser probado.
Ahora, en un artículo publicado en la revista Nature , un equipo dirigido por Mirny y Francois Spitz en el Instituto Pasteur, han demostrado que cohesin realmente desempeña el papel de un motor en el proceso de extrusión de bucles.
“Cada una de estas máquinas cae en el ADN y comienza a extruir bucles, pero hay límites en el ADN que estos motores no pueden pasar”, dice Mirny. “Como resultado de esta actividad motora, el genoma está organizado en muchos bucles dinámicos que no cruzan los límites, por lo que el genoma se divide en una serie de barrios”.
Los investigadores también descubrieron que un mecanismo diferente, que no utiliza cohesin, está trabajando organizando regiones activas e inactivas de ADN en compartimentos separados en el núcleo de la célula.
Para determinar el papel cohesin desempeña en la formación del genoma, el equipo primero suprimió una molécula conocida como Nipbl, que es responsable de la carga cohesin en el ADN.
A continuación, se utilizó una técnica experimental conocida como Hi-C, en la que se capturan y secuencian partes del ADN que están cerca unas de otras en el espacio tridimensional, con el fin de medir la frecuencia de interacciones físicas entre diferentes puntos a lo largo de los cromosomas.
Esta técnica, que fue iniciada por Job Dekker, profesor de bioquímica y farmacología molecular en el Centro Médico de la Universidad de Massachusetts en Worcester, se ha utilizado previamente para demostrar la existencia de TAD.
El equipo utilizó por primera vez la técnica Hi-C para evaluar la organización de los cromosomas antes de extraer la molécula Nipbl de los ratones. A continuación, eliminaron la molécula y realizaron de nuevo la misma medición.
Encontraron que los barrios habían desaparecido virtualmente.
Sin embargo, la compartimentación entre regiones activas e inactivas del genoma se había hecho aún más marcada.
El equipo cree que los motores cohesin permiten a cada gen llegar a sus elementos reguladores, que controlan si los genes deben ser encendidos o apagados.
Es más, parece que los motores de cohesin son detenidos por otra proteína, CTCF, que delimita los límites de cada vecindario. En un estudio reciente en la revista Cell , el laboratorio Mirny, en colaboración con investigadores de la Universidad de California en San Francisco y la Facultad de Medicina de la Universidad de Massachusetts, ha demostrado que si se elimina esta proteína demarcadora, en un vecindario para hablar con elementos reguladores con los que no deberían estar hablando en otro barrio, y conducir a una regulación errónea de los genes en la célula.
“Cohesin es central para la regulación de genes, y enfatizamos que se trata de una función motora, por lo que no es sólo que ellos (genes y sus elementos reguladores) se encuentran en algún lugar aleatorio en el espacio, pero fueron reunidos por esta actividad motora, “Dice Mirny.
Este artículo proporciona nuevas e importantes perspectivas moleculares sobre los mecanismos por los cuales las células doblan sus cromosomas, según Dekker, que no participó en el estudio actual.
“En este trabajo, los laboratorios Mirny y Spitz combinan modelos de ratones con enfoques genómicos para estudiar el plegamiento cromosómico para revelar que la máquina que carga el complejo de cohesina es crítica para la formación de TAD”, dice Dekker. “A partir de este y otro estudio anterior, un mecanismo molecular está entrando en la vista donde TADs forman por cohesin y Nipbl-dependiente de la extrusión de bucles de cromatina, que está bloqueado por sitios vinculados por CTCF”.
Los investigadores ahora están tratando de caracterizar cómo la ausencia del motor molecular afectaría la regulación de genes. También están llevando a cabo simulaciones por ordenador en un intento de determinar cómo la extrusión de bucle basado en cohesina tiene lugar al mismo tiempo que el genoma está experimentando el proceso independiente de segregación en compartimientos activos e inactivos.
“Es como dos pianistas tocando en el mismo piano”, dice Nezar Abdennur, un estudiante de doctorado en el laboratorio de Mirny, que participó en el estudio junto a su compañero de doctorado Anton Goloborodko. “Interfieren y ponen restricciones el uno al otro, pero juntos pueden producir una hermosa pieza de música”.
Abdennur y Goloborodko son co-primeros autores del documento, junto con Wibke Schwarzer de EMBL. La investigación fue apoyada, en parte, por los Institutos Nacionales de la Salud, la Fundación Nacional de la Ciencia y el Fondo MIT-France MISTI.
Fuente: MIT